Pandas里的DataFrame.to_csv()是一个非常常用的函数,用于将内存中的数据以csv格式写到磁盘上,但当要写入的内容较多时,往往会遇到耗时过长的问题。这个问题的原因是to_csv()内部优化不够,我们可以利用其他软件包来曲线的解决此问题。
验证环境
硬件:
- Intel(R) Core(TM) i7-7700HQ CPU @ 2.80GHz 2.81 GHz
- 32.0 GB / 512GB SSD
软件:
- Windows 10 22H2
- Python 3.9.13
- pandas 2.2.2
- numpy 1.26.4
- duckdb 1.0.0
- pyarrow 16.1.0
验证过程
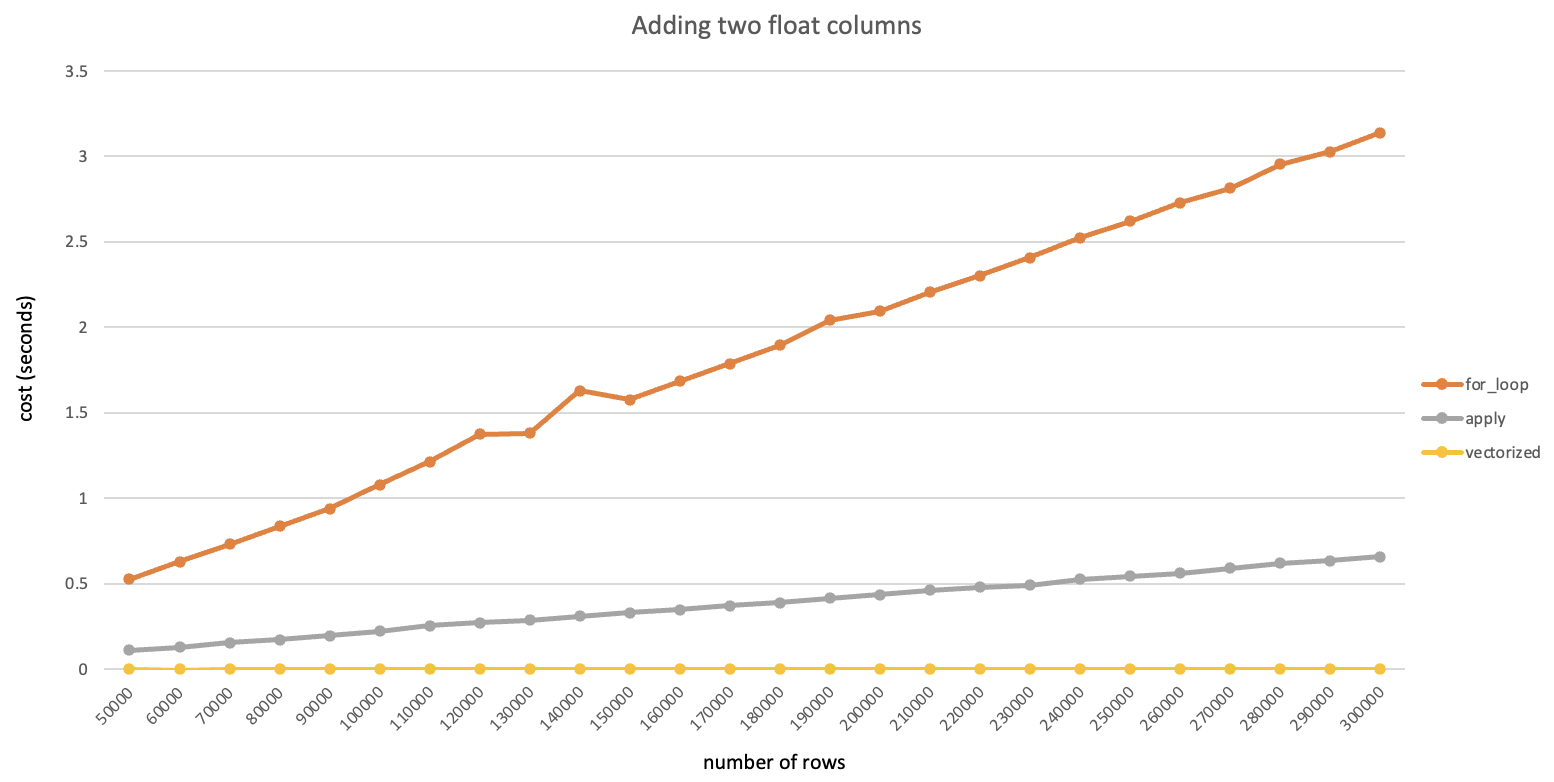
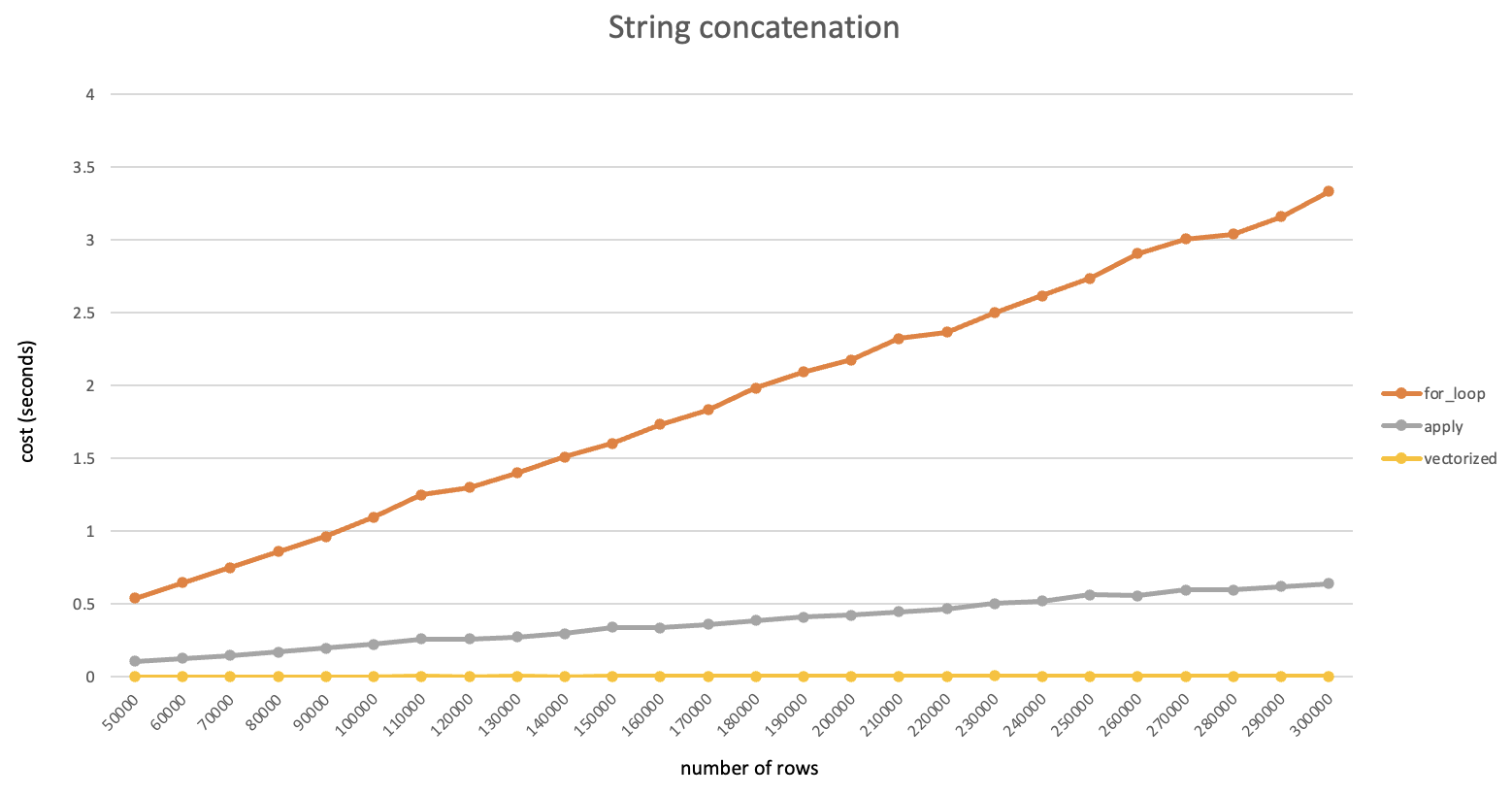
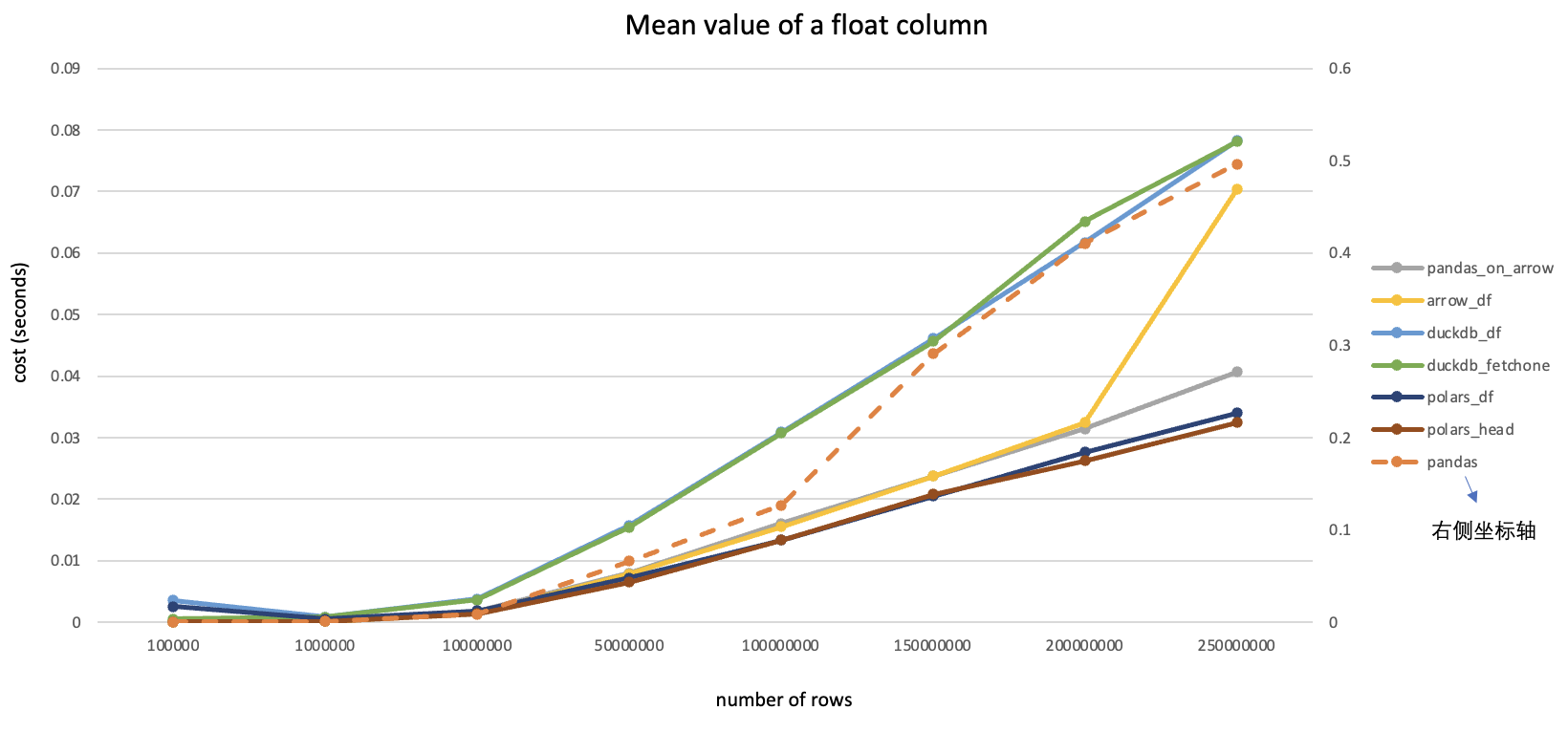
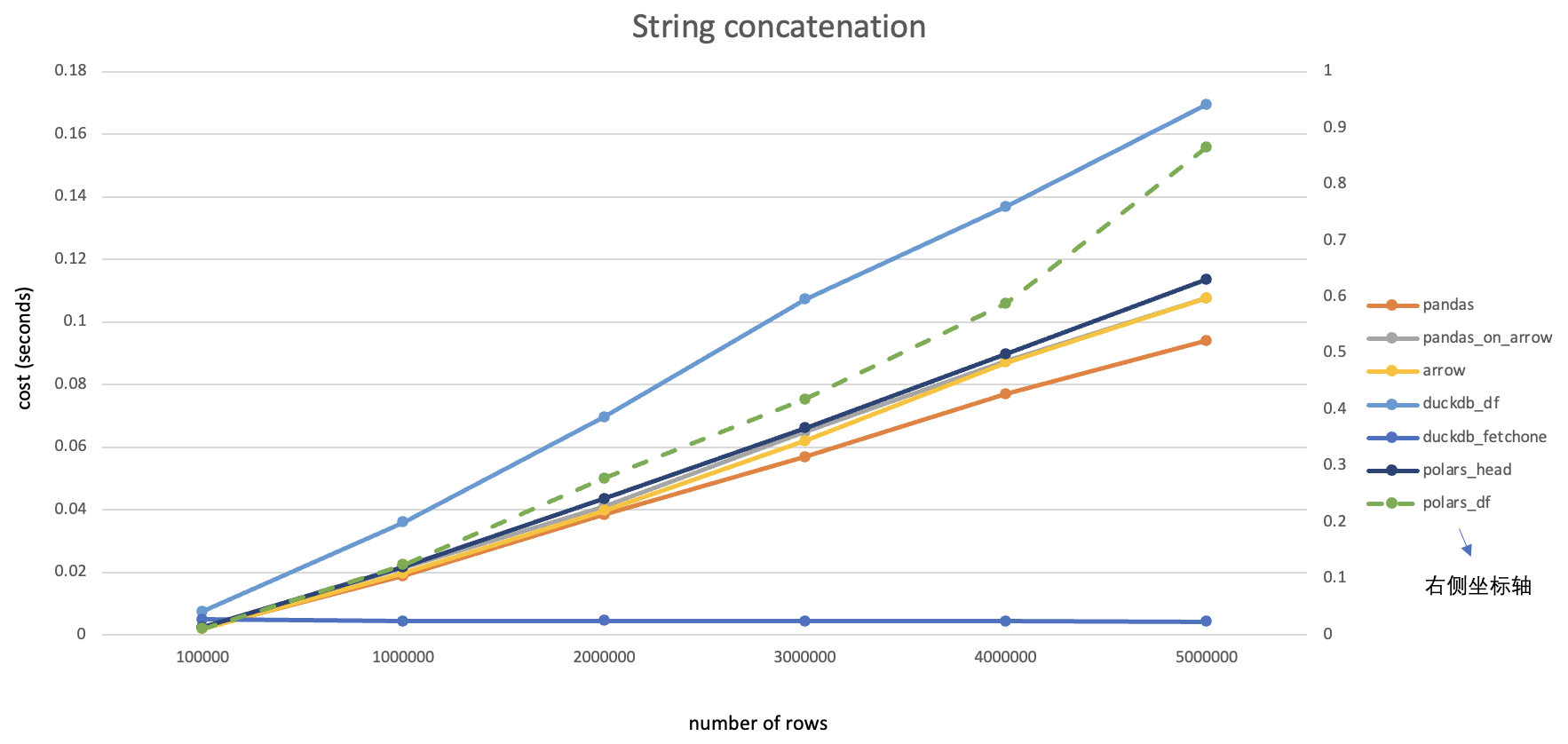
首先随机生成一个较大尺寸的dataframe,内部所有数值都是float64类型,直接调用to_csv()写到磁盘并记录耗时;然后将此dataframe分别转换为numpy、duckdb和pyarrow的对应数据结构(转换过程本身耗时很短可忽略),并统计每次写入csv的耗时(毫秒),整理为下表:
| DataFrame大小 | CSV文件大小 | pandas耗时 | numpy耗时 | duckdb耗时 | pyarrow耗时 |
|---|---|---|---|---|---|
| (10000,100) | 17MB | 1503 | 948 | 529 | 218 |
| (50000,100) | 87MB | 7598 | 4815 | 2683 | 1107 |
| (10000,500) | 87MB | 7341 | 4381 | 2811 | 1085 |
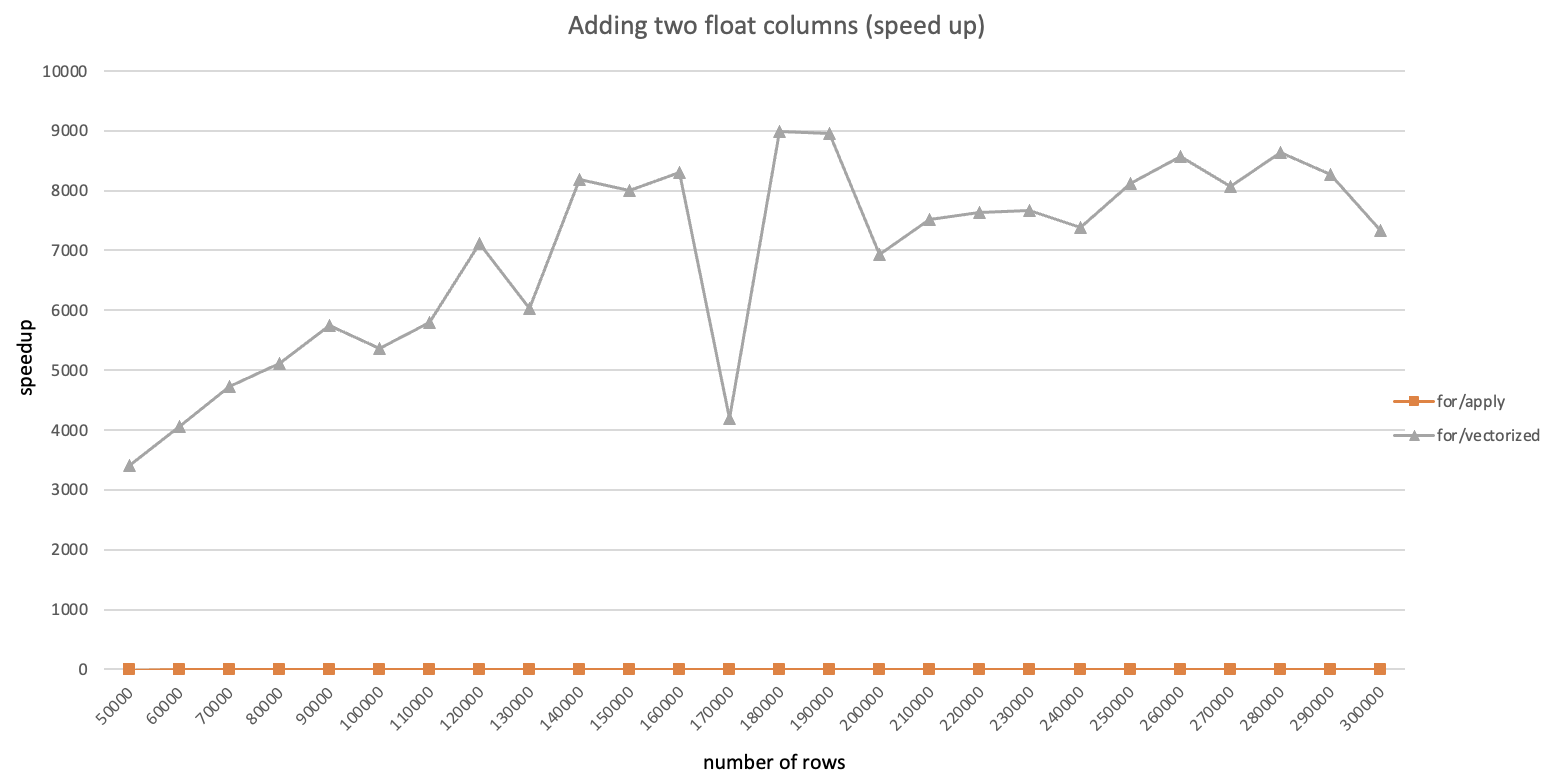
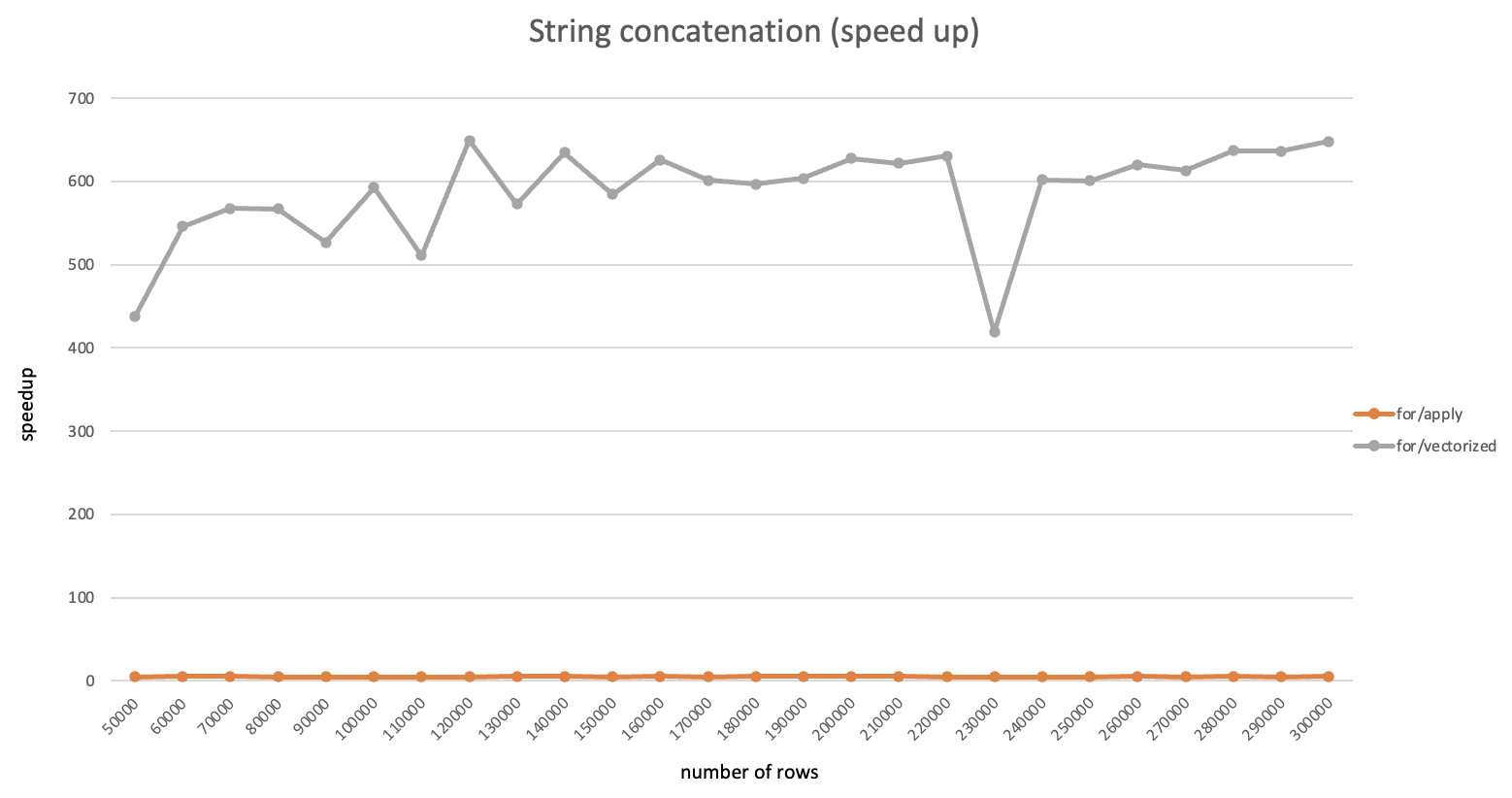
与Pandas.to_csv()对比的性能倍数关系:
| DataFrame大小 | CSV文件大小 | pandas倍数 | numpy倍数 | duckdb倍数 | pyarrow倍数 |
|---|---|---|---|---|---|
| (10000,100) | 17MB | 1 | 1.6 | 2.8 | 6.9 |
| (50000,100) | 87MB | 1 | 1.6 | 2.8 | 6.9 |
| (10000,500) | 87MB | 1 | 1.7 | 2.6 | 6.8 |
注:验证过dask多partition的写入方式,但所得到的不是单个文件因此没有放入表格,并且写入耗时与pandas原生相差不大。
验证结论
可以看到在不同尺寸的数据量下,各软件包的性能都有所提升并且提升幅度比较稳定,其中pyarrow性能最佳,是pandas原生的接近7倍。建议对性能有要求的情况下,将dataframe转换为pyarrow table后再写入。
验证代码:
import pandas as pd
import numpy as np
import time
# 造dataframe数据
np.random.seed(42)
data = np.random.uniform(0, 100, size=(10000, 100))
df = pd.DataFrame(data)
# 直接用to_csv写入文件
# 验证过chunksize, index=False等参数对写入时间影响不大
t0 = time.time() * 1000
df.to_csv('c:/temp/1.csv')
t1 = time.time() * 1000
print(f'pandas cost {t1-t0} ms')
# df转换到numpy后写入csv文件
import numpy as np
numpy_array = df.values
t0 = time.time() * 1000
np.savetxt('c:/temp/1.csv', numpy_array, delimiter=',', fmt='%s')
t1 = time.time() * 1000
print(f'numpy cost {t1-t0} ms')
# df转换到duckdb表后写入csv文件
import duckdb
con = duckdb.connect(database=':memory:', read_only=False)
con.register('my_table', df)
t0 = time.time() * 1000
con.execute("COPY (SELECT * FROM my_table) TO 'c:/temp/2.csv' WITH (FORMAT CSV)")
t1 = time.time() * 1000
print(f'duckdb cost {t1-t0} ms')
# df转换到arrow表后写入csv文件
import pyarrow as pa
import pyarrow.csv as csv
table = pa.Table.from_pandas(df)
t0 = time.time() * 1000
csv.write_csv(table, 'c:/temp/1.csv')
t1 = time.time() * 1000
print(f'pyarrow cost {t1-t0} ms')